Next Generation Hirntumor Gen-Panel Analyse
Genmutationen, d.h. Veränderungen in Genen, spielen eine zentrale Rolle in der Entstehung und der Progression von Tumoren. Man geht davon aus, dass mind. 4 Gene mutiert sein müssen, damit eine Zelle entartet. Die Sequenzierung einer großen Zahl verschiedener Hirntumor-Entitäten hat eine begrenzte Zahl von Genen identifiziert, die häufig bei Hirntumoren mutiert sein können. Diese Erkenntnis liefert neue Möglichkeiten für die individualisierte Diagnostik und Therapie. So sind bestimmte Konstellationen von Mutationen für spezifische Hirntumorentitäten typisch, was eine präzisere Diagnose erlaubt (diagnostische Marker), gehen mit einem besseren klinischen Verlauf einher (prognostische Marker) oder beeinflussen die Wahl der richtigen Therapie (prädiktive Marker). Darüber hinaus können Mutationen Signalmoleküle bzw. -wege betreffen, für welche es bereits spezifische Medikamente in der Klinik oder in der Entwicklung gibt („actionable targets“), so dass sich durch die Identifizierung der Mutationsprofile neue Therapieoptionen ergeben können.
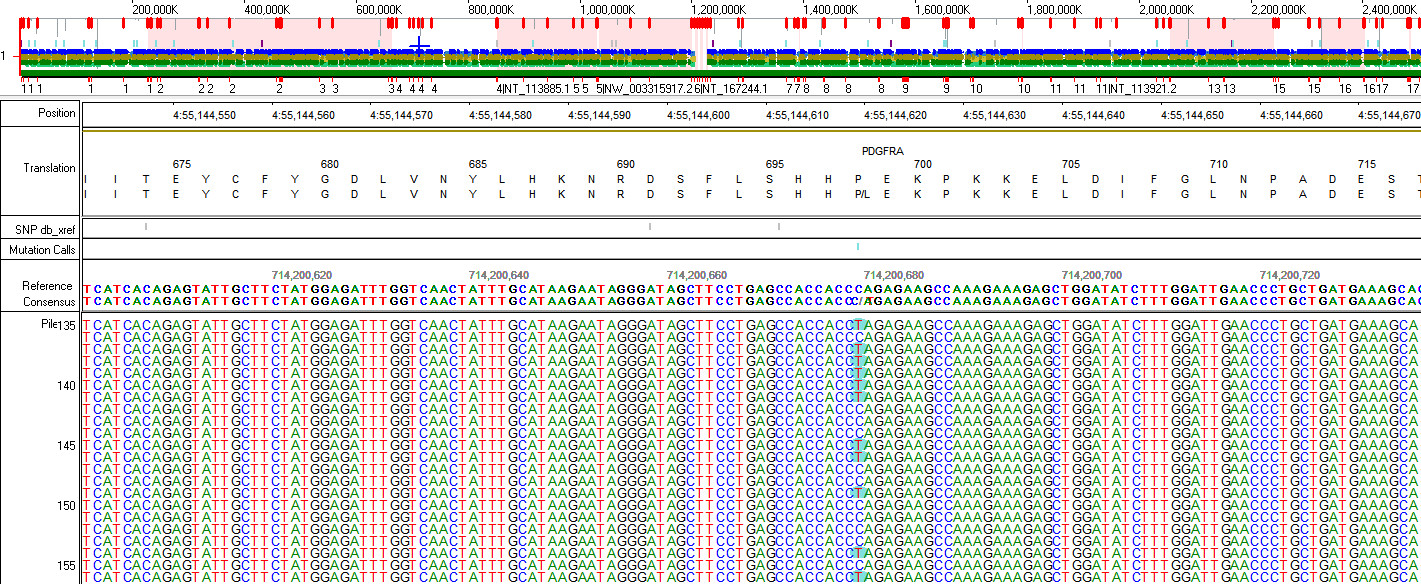
Die Kenntnis über das Mutationsprofil eines Tumors kann somit für die personalisierte Therapie des Patienten von großer Bedeutung sein. Da die Mutationen bei Hirntumoren an vielen verschiedenen Stellen in den betroffenen Genen entstehen können, benötigt ihre Analyse Hochdurchsatz-Methoden für die parallele, d.h. simultane Sequenzierung von Millionen von DNA Fragmenten („next-generation sequencing“). Wir haben zu diesem Zweck einen Hirntumor-Gen-Panel etabliert, welches die wichtigsten und häufigsten in Hirntumoren beobachteten Genmutationen umfasst. Mit diesem Hirntumor-Gen-Panel können simultan Mutationen in 49 Genen über den Illumina MiSeq analysiert werden. Diese Methode ist auch in der Lage, Veränderungen in der Anzahl der Genkopien (Amplifikationen/Deletionen) zu bestimmen. Zur genauen Identifizierung des Tumor-Mutationsprofils über das Hirntumor-Gen-Panel benötigen wir Formalin fixiertes und Paraffin-eingebettetes (FFPE) Material mit repräsentativen Tumoranteilen. Die Einordnung der Befunde erfolgt in enger und interdisziplinärer Zusammenarbeit mit den behandelnden Ärzten.
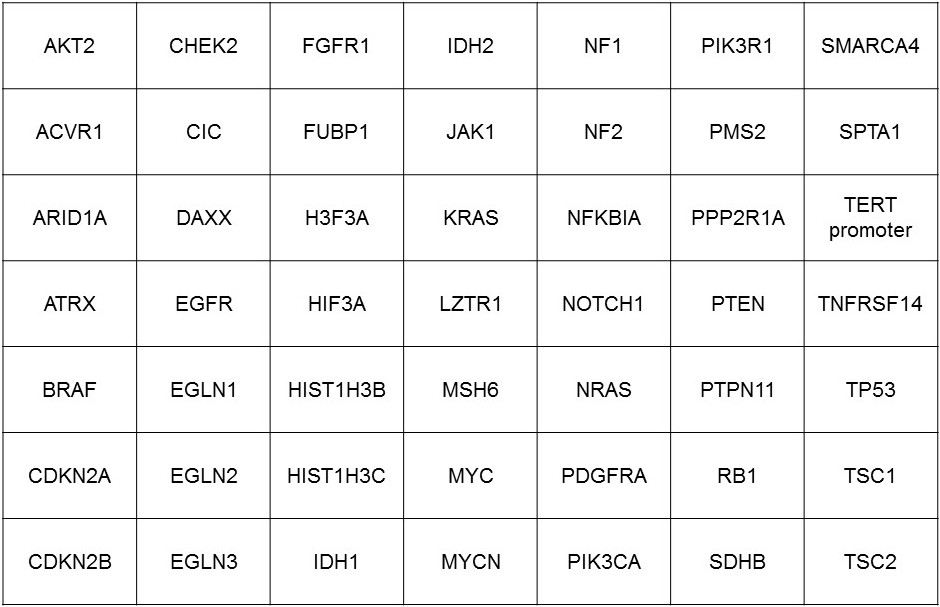
Tabelle der im Hirntumor Gen-Panel eingeschlossenen Gene:
Literaturliste
- Brennan, CW, Verhaak, RG, McKenna, A, Campos, B, Noushmehr, H, Salama, SR, Zheng, S, Chakravarty, D, Sanborn, JZ, Berman, SH, Beroukhim, R, Bernard, B, Wu, CJ, Genovese, G, Shmulevich, I, Barnholtz-Sloan, J, Zou, L, Vegesna, R, Shukla, SA, Ciriello, G, Yung, WK, Zhang, W, Sougnez, C, Mikkelsen, T, Aldape, K, Bigner, DD, Van Meir, EG, Prados, M, Sloan, A, Black, KL et al. The somatic genomic landscape of glioblastoma. Cell 2013; 155(2):462-77
- Cancer Genome Atlas Research Network. Comprehensive, Integrative Genomic Analysis of Diffuse Lower-Grade Gliomas. N Engl J Med 2015; 372(26):2481-98
- Ceccarelli, M, Barthel, FP, Malta, TM, Sabedot, TS, Salama, SR, Murray, BA, Morozova, O, Newton, Y, Radenbaugh, A, Pagnotta, SM, Anjum, S, Wang, J, Manyam, G, Zoppoli, P, Ling, S, Rao, AA, Grifford, M, Cherniack, AD, Zhang, H, Poisson, L, Carlotti, CG, Jr., Tirapelli, DP, Rao, A, Mikkelsen, T, Lau, CC, Yung, WK, Rabadan, R, Huse, J, Brat, DJ, Lehman, NL et al. Molecular Profiling Reveals Biologically Discrete Subsets and Pathways of Progression in Diffuse Glioma. Cell 2016; 164(3):550-63
- Eckel-Passow JE, Lachance DH, Molinaro AM, Walsh KM, Decker PA, Sicotte H, Pekmezci M, Rice T, Kosel ML, Smirnov IV, Sarkar G, Caron AA, Kollmeyer TM, Praska CE, Chada AR, Halder C, Hansen HM, McCoy LS, Bracci PM, Marshall R, Zheng S, Reis GF, Pico AR, O'Neill BP, Buckner JC, Giannini C, Huse JT, Perry A, Tihan T, Berger MS, Chang SM, Prados MD, Wiemels J, Wiencke JK, Wrensch MR, Jenkins RB. Glioma Groups Based on 1p/19q, IDH, and TERT Promoter Mutations in Tumors. N Engl J Med 2015; 372(26):2499-508.
- Sahm, F, Schrimpf, D, Jones, DT, Meyer, J, Kratz, A, Reuss, D, Capper, D, Koelsche, C, Korshunov, A, Wiestler, B, Buchhalter, I, Milde, T, Selt, F, Sturm, D, Kool, M, Hummel, M, Bewerunge-Hudler, M, Mawrin, C, Schuller, U, Jungk, C, Wick, A, Witt, O, Platten, M, Herold-Mende, C, Unterberg, A, Pfister, SM, Wick, W, von Deimling, A. Next-generation sequencing in routine brain tumor diagnostics enables an integrated diagnosis and identifies actionable targets. Acta Neuropathol 2016; 131(6):903-10
- Suzuki, H, Aoki, K, Chiba, K, Sato, Y, Shiozawa, Y, Shiraishi, Y, Shimamura, T, Niida, A, Motomura, K, Ohka, F, Yamamoto, T, Tanahashi, K, Ranjit, M, Wakabayashi, T, Yoshizato, T, Kataoka, K, Yoshida, K, Nagata, Y, Sato-Otsubo, A, Tanaka, H, Sanada, M, Kondo, Y, Nakamura, H, Mizoguchi, M, Abe, T, Muragaki, Y, Watanabe, R, Ito, I, Miyano, S, Natsume, A et al. Mutational landscape and clonal architecture in grade II and III gliomas. Nat Genet 2015; 47(5):458-68